En el diagnóstico de la gripe, resulta fundamental el aislamiento o detección del virus ya que representa una ayuda primordial para el manejo del paciente y para el control de los brotes epidémicos anuales 1. El diagnóstico virológico se fundamenta en dos estrategias:
– Los métodos directos, bien sea la recuperación del virus mediante su aislamiento en cultivo celular o los que permiten detectar la presencia del virus en las secreciones respiratorias del paciente (detección de antígenos o de ácidos nucleicos).
– El diagnóstico indirecto, demuestra y confirma la inducción de una respuesta inmunitaria adaptativa de tipo humoral a través de la detección de anticuerpos específicos en sueros pareados al inicio de la infección y 10-15 días después.
Durante la pandemia del virus gripal H1N1 del 2009, el test recomendado para el diagnóstico, debido a su rapidez y precisión, fue la detección de ARN del virus de la gripe por RT-PCR a tiempo real.
1. Recogida y transporte de las muestras
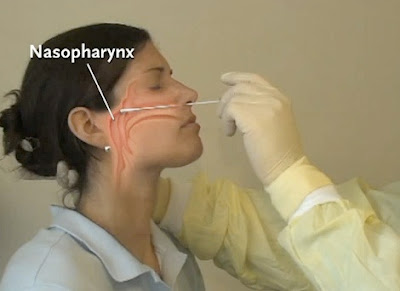
El requisito fundamental a la hora de valorar las muestras obtenidas a partir del tracto respiratorio es que éstas deben contener el mayor número posible de células epiteliales, que son en las que fundamentalmente se replica el virus. Las muestras del tracto respiratorio superior son las más recomendadas para el diagnóstico de la gripe estacional. Para el diagnóstico de H1N1pdm se pueden utilizar muestras de hisopado nasal, hisopado nasofaríngeo, aspirado nasofaríngeo o de garganta. En algunos pacientes hospitalizados se pueden utilizar muestras del tracto respiratorio inferior, como el aspirado bronquial. Estas muestras deben ser tomadas durante los primeros días de la enfermedad.


El transporte de las muestras debe realizarse a 4°C (o en su defecto congeladas a -70°C) con objeto de asegurar la infectividad de las partículas víricas. La recuperación de los virus gripales a partir de la muestra se favorece utilizando un medio de transporte adecuado, que consiste en una solución salina a pH neutro con estabilizadores de proteínas, como el suero de albúmina bovina, y antibióticos para reducir el crecimiento de bacterias que integran la microbiota acompañante.
El procesado de las muestra debe realizarse, bien en laboratorios que tengan niveles de bioseguridad tipo 3 (BSL3) o bien en laboratorios BSL2 con prácticas y elementos de protección de BSL3.
2. Aislamiento mediante cultivo
Inicialmente, el aislamiento de virus requiere medidas de contención de alto nivel de bioseguridad, de acuerdo al documento de la OMS para el tratamiento de este tipo de muestras 2.


Los virus de la gripe son capaces de replicarse en diferentes líneas celulares primarias, diploides o continuas, aunque la susceptibilidad a la infección es baja en la mayoría de ellas. La línea celular más comúnmente utilizada son las células Madin Darby de riñón de perro (MDCK). Las líneas celulares inoculadas con las muestras respiratorias de los pacientes se incuban a 33-35°C en presencia de tripsina para asegurar la activación proteolítica de los virus. La identificación de la existencia de crecimiento del virus sobre la monocapa de células se realiza de modo convencional mediante la observación del efecto citopático causado sobre ellas, que consiste en la aparición de células degenerativas y redondeadas que se desprenden de la monocapa. La caracterización del virus aislado se efectúa por inmunofluorescencia mediante la utilización de anticuerpos monoclonales. En ocasiones el efecto citopático es difícil de apreciar por lo que es necesario disponer de otros métodos para identificar el crecimiento vírico en los cultivos celulares, como son la hemaglutinación, la hemadsorción o la detección de antígenos víricos utilizando técnicas de inmunofluorescencia.
Una de las principales limitaciones del aislamiento de los virus de la gripe es el tiempo necesario de crecimiento e identificación en cultivo celular (4-7 días). Existen varios métodos capaces de detectar la presencia de los virus de la gripe de modo más precoz, entre uno y tres días después de la inoculación de la línea celular y antes de la aparición del efecto citopático. El más comúnmente utilizado es el shell vial, en el que las muestras son directamente centrifugadas sobre la monocapa celular para facilitar la adherencia y penetración vírica. Posteriormente, a las 24h-48h se detecta la presencia de proteínas víricas mediante inmunofluorescencia.
Los virus de la gripe también pueden aislarse tras inocular la muestra en la cavidad alantoidea de huevos de gallina embrionados. Posterior a la inoculación se deben analizar, por hemaglutinación, el fluido amniótico y alantoideo en busca de la presencia de actividad vírica.
3. Detección de antígenos víricos
Los métodos de inmunofluorescencia directa (IFI) y enzimo-inmuno análisis (EIA) se emplean habitualmente para la detección de los antígenos víricos directamente en la muestra clínica o bien en las células del cultivo en las que previamente se ha inoculado la muestra. Los antígenos víricos utilizados generalmente para el diagnóstico son las moléculas que se sitúan en la superficie del virus, la hemaglutinina y la neuraminidasa, que también pueden encontrarse frecuentemente en la superficie de las células infectadas.
4. Diagnóstico molecular
Como ya se ha comentado, el ensayo de RT-PCR a tiempo real fue el método de referencia para el diagnóstico de H1N1pdm. Los CDC pusieron a punto un ensayo que fue autorizado por la FDA (Food and Drug Administration) para su utilización sin restricciones.
5. Detección de la respuesta inmunitaria humoral específica
La identificación indirecta de la infección por los virus de la gripe se realiza mediante ensayos serológicos. Los ensayos más frecuentemente utilizados son la reacción de fijación del complemento, basada en la determinación de anticuerpos frente a la nucleoproteína del virus, la inhibición de la hemaglutinación, basada en la capacidad de la HA viral en hemaglutinar eritrocitos y la neutralización, basada en la capacidad de anticuerpos específicos de inhibir la replicación viral. El fundamento de estas dos últimas técnicas también se detalla en la sección de material y métodos de esta memoria.
6. Secuenciación
La caracterización genética más precisa se realiza mediante la secuenciación de los genes más representativos del genoma viral, seguida de su análisis filogenético y del estudio de aquellos aminoácidos clave implicados en alguna actividad biológica, cuyo cambio puede tener una repercusión clínica y patogénica de la cepa estudiada.
Mediante la secuenciación se puede detectar variaciones que afectaran a la unión del virus a los receptores, cambios en las secuencias génicas que modifican la capacidad antigénica, variaciones que afectan a la actividad enzimática del virus o mutaciones en la secuencias diana de antivíricos, que aporten resistencia al fármaco.
La evaluación de la resistencia a antivirales es determinante para un correcto manejo de la infección.

7. Diagnóstico rápido
Los ensayos de diagnóstico rápido de la gripe basados en la detección de antígenos virales como la NP del virus (proteína altamente conservada) presentan una sensibilidad baja a moderada (10-70%) para el diagnóstico del virus pandémico, de forma que un resultado negativo no significa la ausencia de infección.

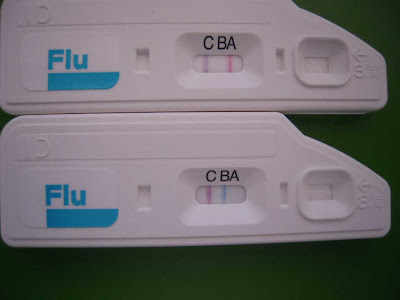
Entre los factores que podrían afectar la sensibilidad, estarían: el tipo y calidad de la muestra, edad del paciente, tiempo transcurrido desde la toma del material hasta su procesamiento y diagnóstico.
El tiempo transcurrido desde la aparición de los síntomas también es un factor clave para el correcto funcionamiento de estos test rápidos, y en general para cualquier test diagnóstico. Se sabe que durante los 3 primeros días de la infección se da un mayor título viral y en consecuencia una mayor sensibilidad.
Bibliografía
1. Eiros JMOdL, R. Tenorio A. et al. Diagnóstico microbiológico de las infecciones víricas respiratorias. Enferm Infecc Microbiol Clin. 2009;27:168-177.
2. Organization WH. WHO Laboratory biorisk management for laboratories handling human specimens suspected or confirmed to contain Influenza A (H1N1) causing the current international epidemics for recommended guidance.
